Polynomiale Regression in R
Die Polynomregression kann als lineare Regression definiert werden, bei der die Beziehung zwischen dem unabhängigen x und dem abhängigen y als Polynom n-ten Grades modelliert wird. Dieses Tutorial zeigt, wie man eine Polynomregression in R durchführt.
Polynomiale Regression in R
Die Polynomregression passt eine nichtlineare Beziehung zwischen x und dem Mittelwert von y an. Es fügt der Regression die polynomischen oder quadratischen Terme hinzu.
Diese Regression wird für eine resultierende Variable und einen Prädiktor verwendet. Die Polynomregression wird hauptsächlich verwendet in:
- Fortschreiten epidemischer Krankheiten
- Berechnung der Wachstumsrate von Geweben
- Verteilung von Kohlenstoffisotopen in Sedimenten
Wir können ggplot2 verwenden, um die Polynomregression in R zu plotten. Wenn dieses Paket noch nicht installiert ist; Zuerst müssen wir es installieren:
install.packages('ggplot2')
Hier ist der schrittweise Prozess der Polynomregression.
Datenerstellung
Wir erstellen einen Datenrahmen mit den Daten der Delftstack-Studenten: die Anzahl der Unterrichtsstunden, die Abschlussprüfungsnoten und die Gesamtzahl der Schüler in der Klasse, die 60 beträgt.
Beispiel:
#create data frame
delftstack <- data.frame(hours = runif(60, 6, 20), marks=60)
delftstack$marks = delftstack$marks + delftstack$hours^3/160 + delftstack$hours*runif(60, 1, 2)
#view the head of the data
head(delftstack)
Dieser Code erstellt die Daten, die später für die Polynomregression verwendet werden.
Ausgang:
hours marks
1 7.106636 71.33509
2 8.501039 74.93339
3 18.051042 124.92229
4 19.153316 141.40656
5 18.306620 118.47464
6 6.240467 70.53522
Datenvisualisierung
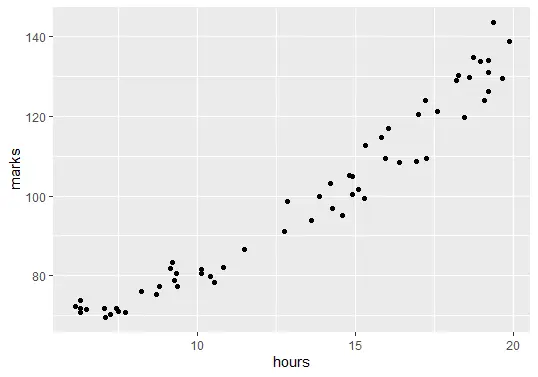
Im nächsten Schritt werden die Daten visualisiert. Bevor wir das Regressionsmodell erstellen, müssen wir den Zusammenhang zwischen der Anzahl der studierten Stunden und den Abschlussnoten aufzeigen.
Beispiel:
# Visualization
library(ggplot2)
ggplot(delftstack, aes(x=hours, y=marks)) + geom_point()
Der obige Code zeichnet das Diagramm unserer Daten:

Passen Sie die polynomialen Regressionsmodelle an
Der nächste Schritt besteht darin, die polynomialen Regressionsmodelle mit den Graden 1 bis 6 und k-facher Kreuzvalidierung mit k = 10 anzupassen.
Beispiel:
#shuffle data
delftstack.shuffled <- delftstack[sample(nrow(df)),]
# number of k-fold cross-validation
K <- 10
#define the degree of polynomials to fit
degree <- 6
# now create k equal-sized folds
fold <- cut(seq(1,nrow(delftstack.shuffled)),breaks=K,labels=FALSE)
#The object to hold MSE's of models
mse_object = matrix(data=NA,nrow=K,ncol=degree)
#K-fold cross validation
for(i in 1:K){
#testing and training data
test_indexes <- which(fold==i,arr.ind=TRUE)
test_data <- delftstack.shuffled[test_indexes, ]
train_data <- delftstack.shuffled[-test_indexes, ]
# using k-fold cv for models evaluation
for (j in 1:degree){
fit.train = lm(marks ~ poly(hours,j), data=train_data)
fit.test = predict(fit.train, newdata=test_data)
mse_object[i,j] = mean((fit.test-test_data$marks)^2)
}
}
# MSE for each degree
colMeans(mse_object)
Ausgang:
[1] 26.13112 15.45428 15.87187 16.88782 18.13103 19.10502
Es gibt sechs Modelle, und die MSEs für jedes Modell sind in der Ausgabe des obigen Codes angegeben. Diese Ausgabe gilt jeweils für die Abschlüsse h=1 bis h=6.
Das Modell mit dem niedrigsten MSE ist unser polynomiales Regressionsmodell, das h = 2 ist, da der MSE-Wert von h = 2 kleiner als alle anderen ist.
Analysieren Sie das endgültige Modell
Lassen Sie uns abschließend das endgültige Modell analysieren und die Zusammenfassung des am besten angepassten Modells zeigen:
Beispiel:
#fitting the best model
best_model = lm(marks ~ poly(hours,2, raw=T), data=delftstack)
#summary of the best model
summary(best_model)
Der obige Code zeigt die Zusammenfassung des am besten geeigneten Modells.
Ausgang:
Call:
lm(formula = marks ~ poly(hours, 2, raw = T), data = delftstack)
Residuals:
Min 1Q Median 3Q Max
-8.797 -2.598 0.337 2.443 9.872
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 68.42847 5.54533 12.340 < 2e-16 ***
poly(hours, 2, raw = T)1 -1.07557 0.93476 -1.151 0.255
poly(hours, 2, raw = T)2 0.22958 0.03577 6.418 2.95e-08 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 4.204 on 57 degrees of freedom
Multiple R-squared: 0.9669, Adjusted R-squared: 0.9657
F-statistic: 831.9 on 2 and 57 DF, p-value: < 2.2e-16
Aus der Ausgabe können wir sehen, dass marks = 68.42847 - 1.07557*(hours) + .22958*(hours)2. Mit dieser Gleichung können wir vorhersagen, wie viele Punkte ein Student basierend auf der Anzahl der studierten Stunden bekommen wird.
Wenn ein Student beispielsweise 5 Stunden studiert hat, lautet die Berechnung:
marks = 68.42847 - 1.07557*(5) + .22958*(5)2
marks = 68.42847 - 1.07557*5 + .22958*25
marks = 68.42847 - 5.37785 + 5.7395
marks = 68.79012
Wenn ein Student 5 Stunden studiert, erhält er in der Abschlussprüfung 68,79012 Punkte.
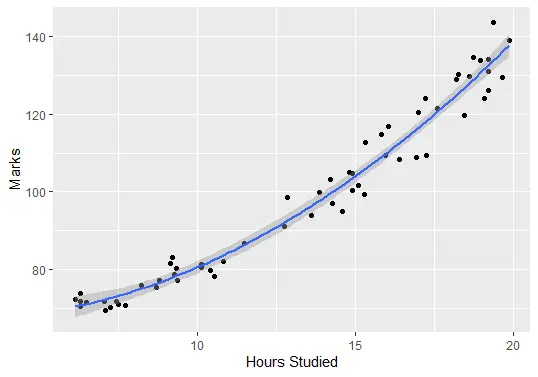
Zuletzt können wir das angepasste Modell plotten, um zu überprüfen, wie gut es den Rohdaten entspricht:
ggplot(delftstack, aes(x=hours, y=marks)) +
geom_point() +
stat_smooth(method='lm', formula = y ~ poly(x,2), size = 1) +
xlab('Hours Studied') +
ylab('Marks')
Ausgabe (Plot):
Sheeraz is a Doctorate fellow in Computer Science at Northwestern Polytechnical University, Xian, China. He has 7 years of Software Development experience in AI, Web, Database, and Desktop technologies. He writes tutorials in Java, PHP, Python, GoLang, R, etc., to help beginners learn the field of Computer Science.
LinkedIn Facebook