Logistische Regression in R
Die logistische Regression ist ein Modell, bei dem die Antwortvariable Werte wie True, False oder 0, 1 hat, die kategoriale Werte sind. Es misst die Wahrscheinlichkeit einer binären Antwort.
Dieses Tutorial zeigt, wie man eine logistische Regression in R durchführt.
Logistische Regression in R
Die Methode glm() wird in R verwendet, um ein Regressionsmodell zu erstellen. Es braucht drei Parameter.
Das erste ist die Formel, das Symbol, das die Beziehung zwischen Variablen darstellt; Zweitens sind die Daten die Datensätze, die die Werte dieser Variablen enthalten; und drittens die Familie, das R-Objekt, das die Details des Modells angibt. Für die logistische Regression ist der Wert binomial.
Der mathematische Ausdruck für die logistische Regression ist unten angegeben:
y = 1/(1+e^-(a+b1x1+b2x2+b3x3+...))
Wo:
aundbsind die numerischen Konstanten, die Koeffizienten sindyist die Antwortvariablexist die Prädiktorvariable
Schritte zum Durchführen einer logistischen Regression in R
Lassen Sie uns nun eine logistische Regression in R durchführen. Hier ist der Schritt-für-Schritt-Prozess.
Laden Sie die Daten
Verwenden wir den Standarddatensatz aus dem ISLR-Paket. Zuerst müssen wir das Paket installieren, falls es noch nicht installiert ist.
install.packages('ISLR')
Sobald das Paket erfolgreich installiert wurde, besteht der nächste Schritt darin, die Daten zu laden.
require(ISLR)
#load the dataset
data_set <- ISLR::Default
## The total observations in data
nrow(data_set)
#the summary of the dataset
summary(data_set)
Der Code lädt den ISLR-Standarddatensatz und zeigt die Anzahl der Beobachtungen und die Datenzusammenfassung an.
Ausgang:
[1] 10000
default student balance income
No :9667 No :7056 Min. : 0.0 Min. : 772
Yes: 333 Yes:2944 1st Qu.: 481.7 1st Qu.:21340
Median : 823.6 Median :34553
Mean : 835.4 Mean :33517
3rd Qu.:1166.3 3rd Qu.:43808
Max. :2654.3 Max. :73554
Der obige Datensatz enthält 10000 Personen.
default zeigt an, ob die Person säumig ist oder nicht, und student gibt an, ob die Person ein Student ist. Der Saldo bezeichnet den durchschnittlichen Saldo einer Person, und das Einkommen ist das Einkommen der Person.
Proben trainieren und testen
Der nächste Schritt besteht darin, den Datensatz in einen Trainings- und einen Testsatz aufzuteilen, um das Modell zu trainieren und zu testen.
#this will make the example reproducible
set.seed(1)
# We use 70% as training and 30% as testing set
sample <- sample(c(TRUE, FALSE), nrow(data), replace=TRUE, prob=c(0.7,0.3))
train <- data_set[sample, ]
test <- data_set[!sample, ]
Erstellen Sie das logistische Regressionsmodell
Wir verwenden glm(), um das logistische Regressionsmodell mit family = binomial zu erstellen.
# the logistic regression model
logistic_model <- glm(default~student+balance+income, family="binomial", data=train)
# just disable scientific notation for summary
options(scipen=999)
# model summary
summary(logistic_model)
Der obige Code erstellt ein logistisches Regressionsmodell und zeigt die Modellzusammenfassung aus den obigen Daten.
Ausgang:
Deviance Residuals:
Min 1Q Median 3Q Max
-2.4881 -0.1327 -0.0509 -0.0176 3.5912
Coefficients:
Estimate Std. Error z value Pr(>|z|)
(Intercept) -10.961385538 0.602044982 -18.207 < 0.0000000000000002 ***
studentYes -0.835485760 0.284855225 -2.933 0.00336 **
balance 0.005893470 0.000289649 20.347 < 0.0000000000000002 ***
income -0.000001611 0.000009942 -0.162 0.87124
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
(Dispersion parameter for binomial family taken to be 1)
Null deviance: 1997.4 on 7014 degrees of freedom
Residual deviance: 1050.9 on 7011 degrees of freedom
AIC: 1058.9
Number of Fisher Scoring iterations: 8
Das logistische Regressionsmodell wurde erfolgreich erstellt. Dann besteht der nächste Schritt darin, das Modell zu verwenden, um Vorhersagen zu treffen.
Verwenden Sie das Modell, um Vorhersagen zu treffen
Sobald das logistische Regressionsmodell angepasst ist, können wir es verwenden, um vorherzusagen, ob die Person auf der Grundlage des Status Einkommen, student oder Saldo in Ausfall geraten wird.
#defining two individuals
demo <- data.frame(balance = 1500, income = 3000, student = c("Yes", "No"))
#predict the probability of defaulting
predict(logistic_model, demo, type="response")
Der obige Code sagt die Ausfallwahrscheinlichkeit von zwei definierten Personen voraus.
Ausgang:
1 2
0.04919576 0.10659389
Die Wahrscheinlichkeit, dass eine Person mit einem Guthaben von $1500, einem Einkommen von $3000 und einem Studentenstatus von ja zahlungsunfähig wird, beträgt 0.0491. Ebenso bei einem Studenten-Status von nein beträgt die default-Wahrscheinlichkeit 0.1065.
Lassen Sie uns nun die Ausfallwahrscheinlichkeit jedes Einzelnen in unserem Testdatensatz berechnen.
#probability of default for the test dataset
prediction <- predict(logistic_model, test, type="response")
Der Code berechnet die Ausfallwahrscheinlichkeit für jede Person in unserem Testdatensatz. Die Ausgabe wird eine große Summe von Daten sein.
Die Diagnose des logistischen Regressionsmodells
Jetzt ist es an der Zeit zu überprüfen, wie gut unser Modell mit dem Testdatensatz abschneidet. Die optimale Wahrscheinlichkeit ermitteln wir mit der Methode optimalCutoff() aus der Bibliothek informationvalue.
Beispiel:
library(InformationValue)
#from "Yes" and "No" to 1's and 0's
test$default <- ifelse(test$default=="Yes", 1, 0)
#optimal cutoff probability to use for maximize accuracy
optimal <- optimalCutoff(test$default, prediction)[1]
optimal
Die zu verwendende optimale Wahrscheinlichkeitsgrenze ist unten angegeben. Jede Person mit einer höheren Wahrscheinlichkeit gilt als ausgefallen.
Ausgang:
[1] 0.5209985
Als nächstes können wir die Konfusionsmatrix verwenden, um den Vergleich unserer Vorhersage mit tatsächlichen Standardwerten zu zeigen.
Beispiel:
confusionMatrix(test$default, prediction)
Ausgang:
0 1
0 2868 71
1 10 36
Wir können auch die True-Positiv-Rate (Sensitivität), die True-Negativ-Rate (Spezifität) und den Fehlklassifizierungsfehler berechnen:
# sensitivity
sensitivity(test$default, prediction)
# specificity
specificity(test$default, prediction)
# total misclassification error rate
misClassError(test$default, prediction, threshold=optimal)
Ausgang:
[1] 0.3364486
[1] 0.9965254
[1] 0.0265
Die Gesamtfehlerrate bei Fehlklassifizierungen beträgt für unser Modell 2,65 %, was bedeutet, dass unser Modell Ergebnisse leicht vorhersagen kann, da die Fehlerrate sehr niedrig ist.
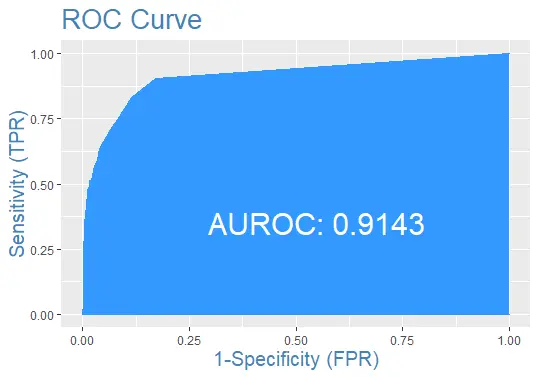
Zeichnen wir abschließend die ROC-Kurve für den Testdatensatz mit der Vorhersage:

Vollständiger Beispielcode
Hier ist der vollständige Code, der in diesem Tutorial verwendet wird.
install.packages('ISLR')
require(ISLR)
#load the dataset
data_set <- ISLR::Default
## The total observations in data
nrow(data_set)
#the summary of the dataset
summary(data_set)
#this will make the example reproducible
set.seed(1)
#We use 70% of as training set and 30% as testing set
sample <- sample(c(TRUE, FALSE), nrow(data), replace=TRUE, prob=c(0.7,0.3))
train <- data_set[sample, ]
test <- data_set[!sample, ]
# the logistic regression model
logistic_model <- glm(default~student+balance+income, family="binomial", data=train)
# just disable scientific notation for summary
options(scipen=999)
#view model summary
summary(logistic_model)
#defining two individuals
demo <- data.frame(balance = 1500, income = 3000, student = c("Yes", "No"))
#predict the probability of defaulting
predict(logistic_model, demo, type="response")
#probability of default for the test dataset
prediction <- predict(logistic_model, test, type="response")
install.packages('InformationValue')
library(InformationValue)
#from "Yes" and "No" to 1's and 0's
test$default <- ifelse(test$default=="Yes", 1, 0)
#optimal cutoff probability to use for maximize accuracy
optimal <- optimalCutoff(test$default, prediction)[1]
optimal
confusionMatrix(test$default, prediction)
# sensitivity
sensitivity(test$default, prediction)
# specificity
specificity(test$default, prediction)
# total misclassification error rate
misClassError(test$default, prediction, threshold=optimal)
#the ROC curve
plotROC(test$default, prediction)
Sheeraz is a Doctorate fellow in Computer Science at Northwestern Polytechnical University, Xian, China. He has 7 years of Software Development experience in AI, Web, Database, and Desktop technologies. He writes tutorials in Java, PHP, Python, GoLang, R, etc., to help beginners learn the field of Computer Science.
LinkedIn Facebook