Comment fixer les limites des axes dans les tracés ggplot2 R
-
Fixez les limites de l’axe des x avec
scale_x_continuousdansggplot2 -
Utilisez
coord_cartesianpour limiter les deux axes dansggplot2
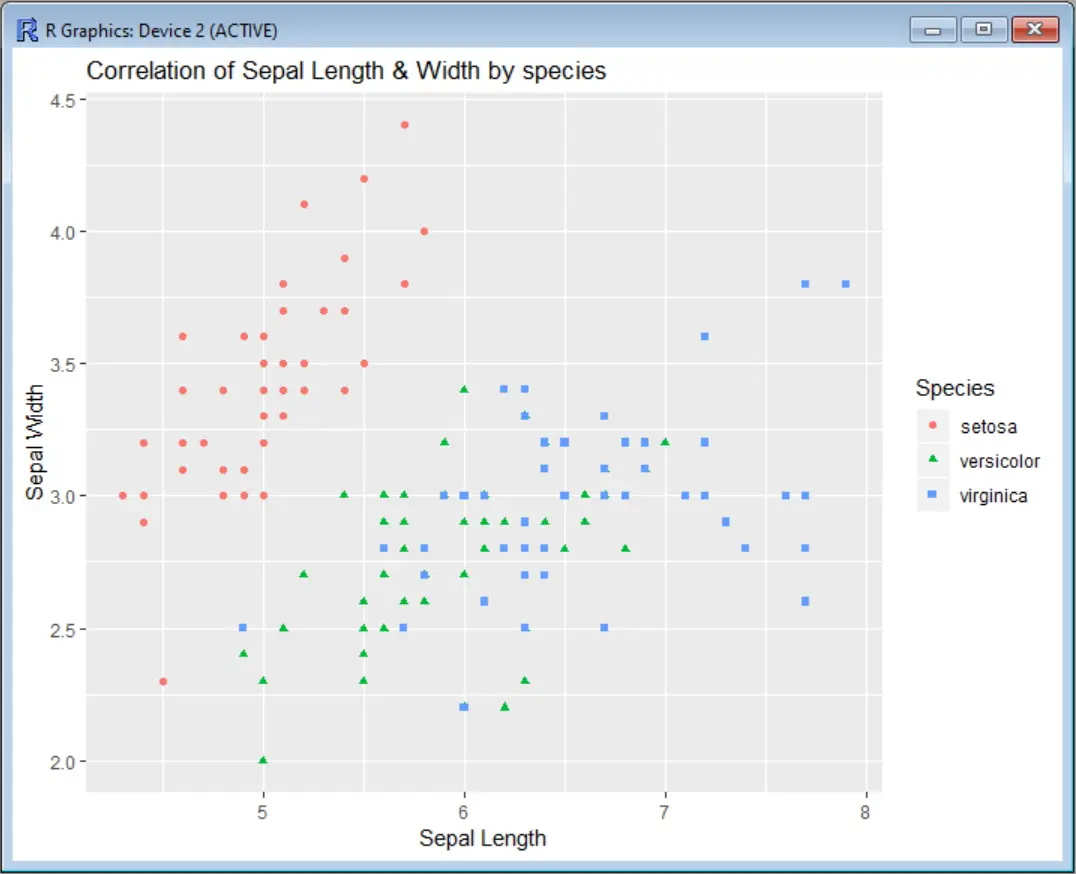
Voyons d’abord notre diagramme de dispersion sans mettre de limite à l’axe. Dans cet exemple, nous utilisons l’ensemble de données Iris pour corréler les valeurs de largeur des sépales et la largeur des sépales, en classant les espèces par couleur. Avant d’appeler la fonction ggplot, nous devons installer le paquet ggplot2 et charger la bibliothèque correspondante :
install.packages("ggplot2")
library(ggplot2)
scatter <- ggplot(data=iris, aes(x = Sepal.Length, y = Sepal.Width))
scatter + geom_point(aes(color=Species, shape=Species)) +
ylab("Sepal Width") + xlab("Sepal Length") +
ggtitle("Correlation of Sepal Length & Width by species")
Output :

Maintenant, supposons que nous voulions limiter l’axe des x (longueur des sépales) de sorte que le graphique ne montre que la région comprise entre x=5 et x=7. Nous avons deux options pour cela : utiliser scale_x_continuous ou utiliser coord_cartesian.
L’option scale_x_continuous supprime tous les points de données qui se trouvent en dehors de la plage spécifiée pour l’axe donné, tandis que l’option coord_cartesian n’ajuste que la zone visible. Dans la plupart des cas, les deux options donneront les mêmes résultats. Mais si vous ajustez quelque chose aux données, les valeurs ajustées changeront probablement.
Fixez les limites de l’axe des x avec scale_x_continuous dans ggplot2
Pour utiliser scale_x_continuous, vous devez spécifier un vecteur avec les limites inférieure et supérieure de l’axe des x, comme ceci :
scatter <- ggplot(data=iris, aes(x = Sepal.Length, y = Sepal.Width))
scatter + geom_point(aes(color=Species, shape=Species)) +
ylab("Sepal Width") + xlab("Sepal Length") +
ggtitle("Correlation of Sepal Length & Width by species") +
scale_x_continuous(limits = c(5, 7))
Production :
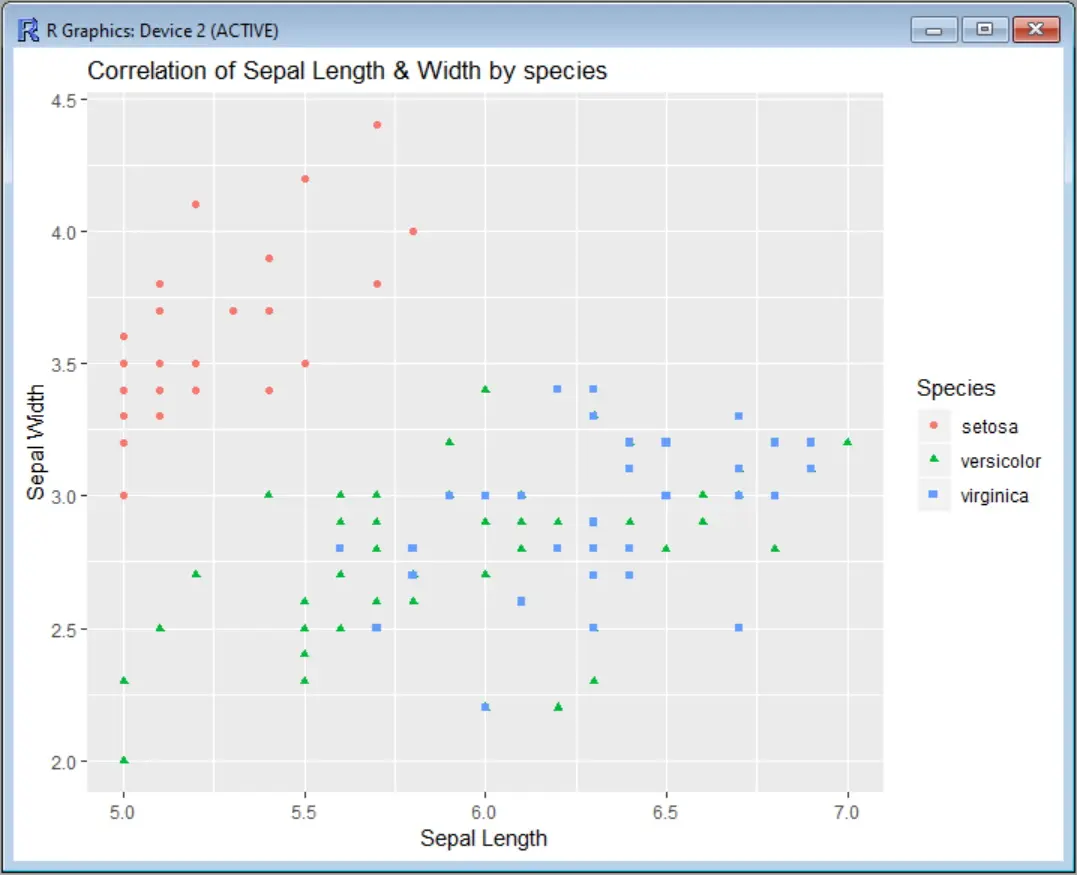
Vous recevrez également un message d’avertissement vous indiquant combien de points de données sont éliminés du graphique :
Warning message:
Removed 34 rows containing missing values (geom_point).
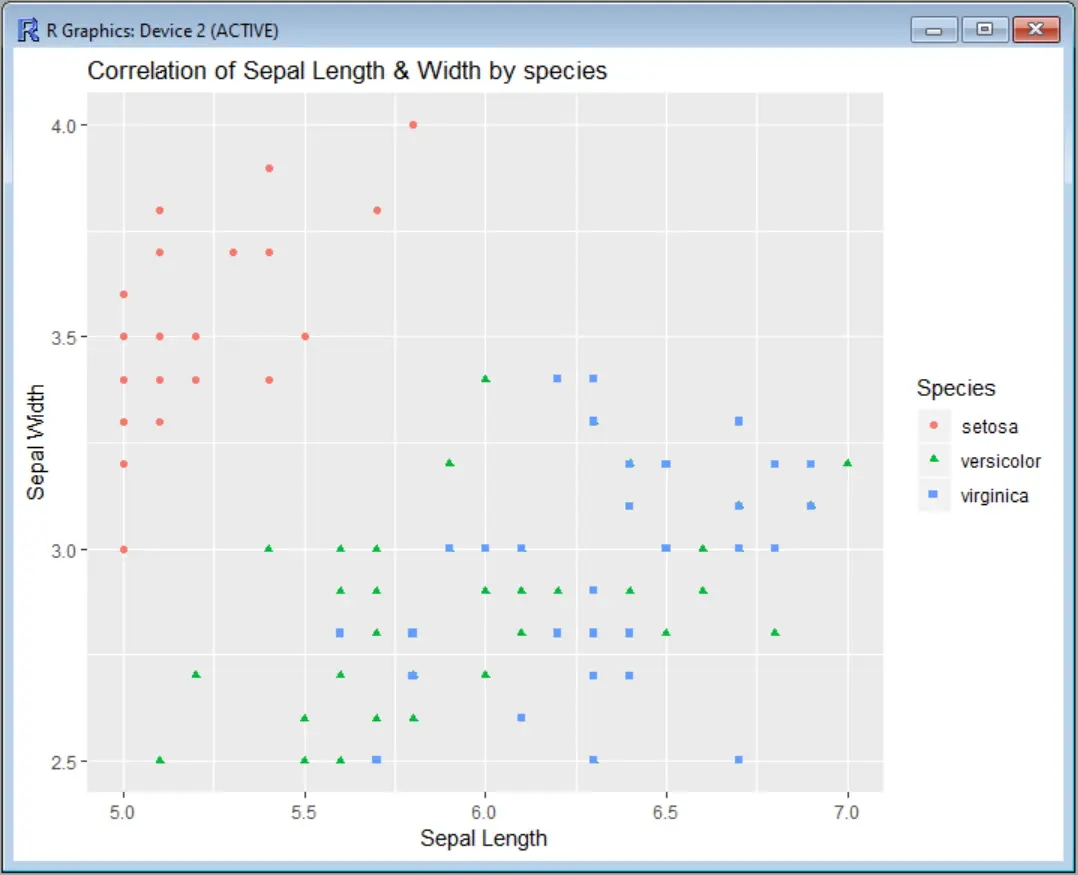
Si vous souhaitez également limiter l’axe des y, vous pouvez ajouter scale_y_continuous de la même manière, comme dans cet exemple :
scatter-<--ggplot(data=iris,-aes(x-=-Sepal.Length,-y-=-Sepal.Width))
scatter + geom_point(aes(color=Species, shape=Species)) +
ylab("Sepal Width") + xlab("Sepal Length") +
ggtitle("Correlation of Sepal Length & Width by species") +
scale_x_continuous(limits = c(5, 7)) +
scale_y_continuous(limits = c(2.5, 4))
La sortie affichera les limites sur les deux axes :
Vous pouvez également utiliser les fonctions d’abréviations xlim et ylim au lieu de scale_x_continuous et scale_y_continuous pour rendre la commande un peu plus courte :
scatter <- ggplot(data=iris, aes(x = Sepal.Length, y = Sepal.Width))
scatter + geom_point(aes(color=Species, shape=Species)) +
ylab("Sepal Width") + xlab("Sepal Length") +
ggtitle("Correlation of Sepal Length & Width by species") +
xlim(5, 7) + ylim(2.5, 4)
Utilisez coord_cartesian pour limiter les deux axes dans ggplot2
Si vous voulez utiliser coord_cartesian au lieu de scale_x_continuous et scale_y_continuous, la commande serait comme ceci :
scatter <- ggplot(data=iris, aes(x = Sepal.Length, y = Sepal.Width))
scatter + geom_point(aes(color=Species, shape=Species)) +
ylab("Sepal Width") + xlab("Sepal Length") +
ggtitle("Correlation of Sepal Length & Width by species") +
coord_cartesian(xlim = c(5, 7), ylim = c(2.5, 4))
Et la sortie du tracé sera la même.
Dans le RStudio cheatsheet de ggplot2, vous pouvez trouver une explication visuelle pour l’utilisation de toutes les options de cette commande.